Actualités
UE – OMS contre AESA : des experts mondiaux s’affrontent sur les gènes de résistance aux antibiotiques dans les PGM
L’Organisation Mondiale de la Santé (OMS) et l’Agence Européenne des Sécurité Alimentaire (AESA) divergent indirectement sur l’utilisation des gènes de résistance à des antibiotiques. Certains dossiers, à l’image de la pomme de terre Amflora de BASF [1], ont en effet pour point de discorde – parmi d’autres – la présence d’un tel gène. Ces gènes de résistance sont utilisés comme gènes marqueurs permettant de sélectionner les cellules ayant bien intégré le transgène d’intérêt agronomique. Or un tel usage peut conduire à une dissémination de la résistance aux antibiotiques ce qui, dans le domaine médical par exemple, pourrait s’avérer très problématique. L’Organisation Mondiale de la Santé (OMS) a d’ailleurs pris position sur le sujet en 2008, en rangeant les antibiotiques dans plusieurs catégories selon leur « importance » médicale et en considérant bien sûr que les plus importants ne sauraient être utilisés dans des domaines autres que médicaux afin de préserver ces médicaments [2].
En Europe, l’Agence Européenne de Sécurité Alimentaire (AESA) avait déjà publié une opinion en 2004 sur l’utilisation de ces gènes de résistance dans laquelle elle expliquait avoir classifié les gènes en trois groupes [3], à l’instar de l’OMS, selon l’utilisation médicale des antibiotiques correspondants. Mais si la démarche est la même, AESA et OMS ne sont pas d’accord sur les résultats de la classification. Ainsi, on observe que si pour l’AESA, les gènes de résistance à la streptomycine et l’ampicilline sont utilisables dans des PGM (même commercialement), il s’agit pour l’OMS d’antibiotiques d’une importance critique en médecine, donc à ne jamais utiliser ailleurs. En ce qui concerne la kanamycine, la néomycine et la spectinomycine, il s’agit d’antibiotiques d’une importance forte pour l’OMS, donc d’utilisation à restreindre ailleurs.
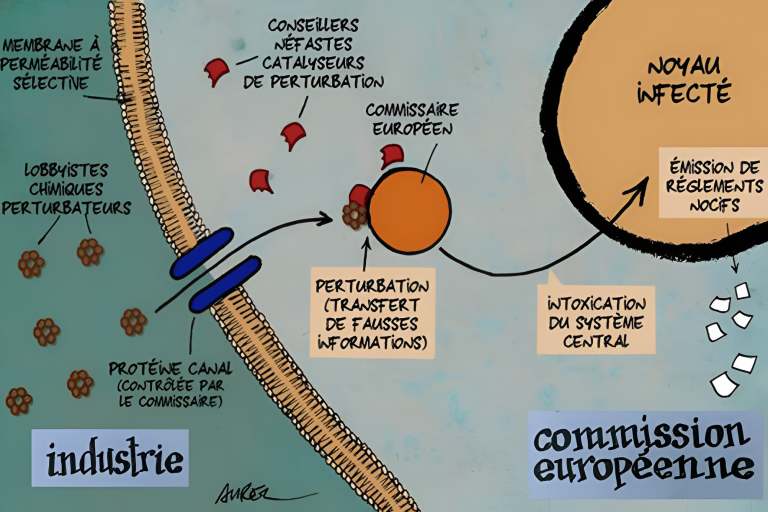
Suite aux désaccords sur la pomme de terre Amflora, la Commission européenne a donc demandé à l’AESA de revoir son opinion afin de la clarifier et de mieux définir la procédure d’autorisations des PGM. Dans cette « nouvelle opinion » [4], l’AESA affirme que son avis précédent répond déjà aux exigences d’évaluation et qu’aucune nouvelle donnée scientifique n’étant apparue, point n’est besoin de réactualiser cette opinion. L’AESA explique également que le transfert de gène de résistance d’une PGM à une bactérie n’a pas été démontré, que le transfert d’ADN à des bactéries est de faible fréquence et que la résistance à la kanamycine ou la néomycine est déjà très répandue dans l’environnement. Sur la différence de point de vue avec l’OMS, l’AESA explique simplement que le gène nptII n’est pas impliqué dans les cas de tuberculoses multirésistantes aux antibiotiques, signifiant ainsi qu’il n’est pas problématique de l’utiliser. Pour l’AESA donc, les gènes de résistance aux antibiotiques, c’est (presque) automatique dans les PGM…
[3] http://www.efsa.europa.eu/EFSA/efsa…
Groupe 1 : nptII (kanamycine, néomycine…) et hph (hygromycine), utilisation dans les PGM permise sans restriction
Groupe 2 : CmR (chloramphenicol), ampr (ampicilline), aadA (streptomycine et spectinomycine), utilisation seulement pour les essais en champs
Groupe 3 : nptIII (amikacine) et TetA (tetracyclines), interdits dans les PGM.